- Abteilungen
- Abteilung Molekulare Ökologie
Abteilung Molekulare Ökologie
Gruppen
Motivation und Fokus unserer Abteilung
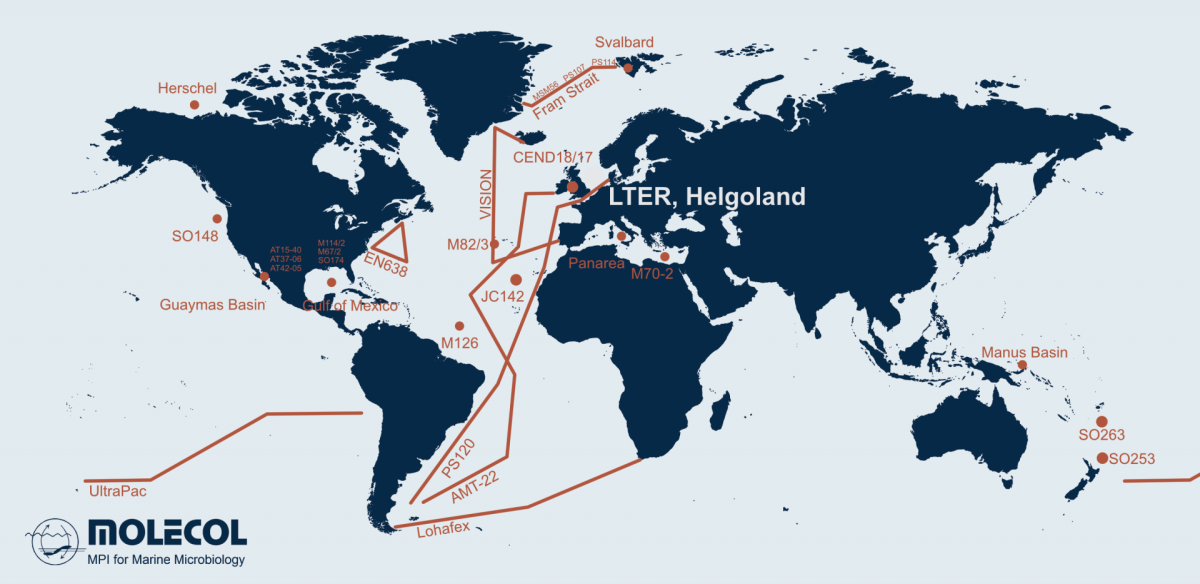
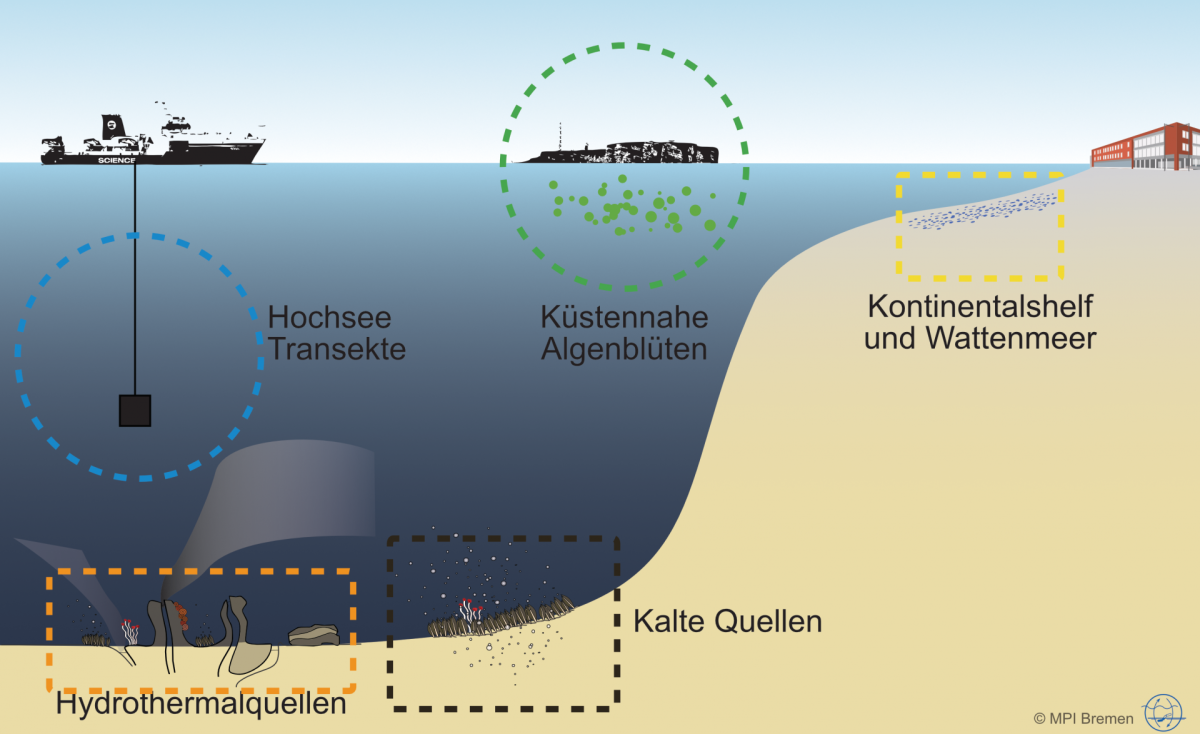
Mikroorganismen sind verantwortlich für etwa 70 % der Gesamtbiomasse im Meer und sie sind unabdingbar für das Ökosystem Meer. Sie regulieren die Vorkommen von organischer Materie, leiten globale Stoffkreisläufe und beeinflussen das Weltklima. Obwohl tausende Bakterien- und Archaeen-Arten isoliert und beschrieben wurden, finden wir Millionen mit molekularbiologischen Methoden in der Umwelt. Unsere Abteilung kombiniert modernste Techniken in einem fachübergreifenden Ansatz, um die Ökologie von mikrobiellen Gemeinschaften zu erforschen; und das von Hydrothermalquellen bis zum Oberflächenwasser und von den Tropen bis in Polarregionen.
Projekte
Geschäftsführender Direktor
MPI für Marine Mikrobiologie
Celsiusstr. 1
D-28359 Bremen
|
Raum: |
2221 |
|
Telefon: |

Assistentin
MPI für Marine Mikrobiologie
Celsiusstr. 1
D-28359 Bremen
|
Raum: |
1345 |
|
Telefon: |

News
"Wir gratulieren Isidora Morel zur erfolgreichen Verteidigung ihrer Doktorarbeit"
[17.11.2025]
Auch unkultivierte Mikroben brauchen eine eigene Taxonomie
Die Taxonomie umfasst die Identifizierung, Klassifizierung und Nomenklatur von Organismen. Als solche ist die Taxonomie eine Voraussetzung für die Studien der Ökologie. Nur auf der Grundlage genauer taxonomischer Konzepte und Methoden kann die Vielfalt und Zusammensetzung komplexer mikrobieller Gemeinschaften genau beschrieben und überwacht werden. Unsere Abteilung blickt auf eine lange Tradition in der Entwicklung und Anwendung neuer taxonomischer Methoden zurück. Dabei haben wir Pionierarbeit bei der in-situ-Identifizierung, Quantifizierung und Lokalisierung von bislang unkultivierten Bakterien und Archaeen mittels Fluoreszenz-in-situ-Hybridisierung (FISH) geleistet und wesentlich zu einer standardisierten Klassifizierung auf 16S rRNA Grundlage beigetragen.
Die Fortschritte in der DNA-Sequenzierung in den letzten Jahren versorgen uns mit immer mehr Informationen über noch unkultivierte Mikroorganismen. Durch die geschickte Kombination verschiedener Methoden, wie Metagenomic und FISH, haben wir in den letzten Jahren mehrere funktionell wichtige Taxa beschreiben können. Dazu zählen zum Beispiel die Art Candidatus Prosiliicoccus vernus (Francis et al. 2019) und die Gattung Candidatus Abditibacter (Grieb et al. 2020). Finde mehr heraus über unsere Arbeit mit der Taxonomie unkultivierter Bakterien und Archaeen auf unserer Projektseite Taxonomie.
Diversität, Visualisierung und Kultivierung
„Wer oder was ist da?" ist eine der Hauptfragen in der Ökologie. In der marinen mikrobiellen Ökologie sequenzieren wir typischerweise das 16S rRNA-Gen mit modernsten Sequenzierungsmethoden, um einen Einblick in die Vielfalt der mikrobiellen Gemeinschaft zu erhalten. So können wir die wichtigsten mikrobiellen Taxa identifizieren und Hypothesen über ökologische Zusammenhänge formulieren. Die Entwicklung der Sequenzierungstechnologien der letzten Jahre hat die Einführung der PacBio Sequell II-Plattform hervorgebracht. Diese Plattform sequenziert besonders lange DNA-Sequenzen, so dass wir nun das gesamte genetische Material von Umweltpopulationen sequenzieren (Metagenomik) und gleichzeitig die 16S rRNA-Gene in voller Länge gewinnen können. Im Vergleich zu den deutlich kürzeren DNA-Sequenzen herkömmlicher Methoden können die längeren Sequenzen für robustere und genauere phylogenetische Analysen verwendet werden.
Die nächste grundlegende Komponente der Ökologie ist die Beurteilung der Abundanz einer Population in der Umwelt. Vereinfacht können wir sagen: Je mehr Individuen einer Art vorhanden sind, desto wichtiger ist sie für das Ökosystem und desto größer ist ihr Einfluss auf den Nährstoffkreislauf, die Ressourcenexploration und metabolische Interaktionen. Hier ist unsere Methode der Fluoreszenz in-situ-Hybridisierung (FISH) ein unverzichtbares Werkzeug für die Zählung taxonomisch genau definierter Mikroorganismen.
Da sich die meisten Mikroben in ihrer Morphologie stark ähneln und sie sich somit nicht differenzieren lassen, ist der einzige verlässliche Marker zur Unterscheidung das ribosomale RNA-Molekül, das hoch konserviert und in jedem Organismus reichlich vorhanden ist. Wir können kleine fluoreszenzmarkierte DNA-Fragmente, die komplementär zur rRNA sind, für die wichtigsten mikrobiellen Gruppen maßschneidern und sie mittels Fluoreszenzmikroskopie sichtbar machen so wie zählen. Neben einer numerischen Quantifizierung liefert FISH einen Hinweis auf zelluläre Strukturen und physikalische Interaktionen mit anderen Mikroben. Mit Hilfe von hochauflösender Mikroskopie, wie der konfokalen Laser Scanning Mikroskopie (CLSM) und der STimulated Emission Depletion (STED) Mikroskopie, können wir intrazelluläre Eigenschaften oder die Aufnahme von z. B. Substraten untersuchen. Mit Hilfe von FISH und hochauflösender Mikroskopie können wir genaue Zellgrößen und Biovolumina von unkultivierten Mikroben bestimmen.
Obwohl wir aus Umweltproben direkt eine Fülle von Informationen über die Ökologie ihrer Mikroorganismen gewinnen können, sind wir für ein vollständiges Verständnis der Funktionen und ökologischen Zusammenhänge von Reinkulturen oder Anreicherungen im Labor abhängig. Die Kultivierung ist in den meisten Fällen noch immer die einzige Möglichkeit, die Vorhersagen und Hypothesen, die aus genomischen Sequenzinformationen formuliert wurden, experimentell zu verifizieren. Die Isolierung von marinen Mikroorganismen ist jedoch nicht trivial, da es eine Herausforderung ist, die natürliche Umgebung und die spezifischen Bedingungen, die für das Wachstum einer Population erforderlich sind, nachzubilden. Wir verwenden spezifische Flüssigmedien und Anreicherungen zusammen mit der physischen Trennung einzelner Zellen durch Verdünnung und molekulare Identifizierungsmethoden (im Haus entwickelte spezifische PCR- und FISH), um abundante Mikroben aus natürlichen Gemeinschaften zu isolieren. Sobald ein Stamm in Reinkultur vorliegt, ist für ausreichend Nachschub gesorgt. Wir können dann physiologische Merkmale und individuelle Enzyme untersuchen.

Funktionelle Charakterisierung
Die rasante Entwicklung neuer Sequenzierungstechnologien hat eine neue Ära für die Untersuchung der genetischen Möglichkeiten mikrobieller Gemeinschaften eingeläutet. Von der Analyse sequenzierter Populationen in der Natur bis hin zu Isolaten im Labor erlaubt uns der Einsatz von Multi-omic-Techniken die Integration genetischer Informationen auf Gen-, Transkript- und auch Proteinebene. Unser Ziel ist es, die Lücke zwischen vorhergesagter Funktion und gemessener mikrobieller Aktivität durch die Integration dieser verschiedenen Informationsebenen zu schließen.
Die mRNA von Spezies innerhalb mikrobieller Gemeinschaften verrät uns, welche Gene aktiv transkribiert werden, welches wir mit einem (Meta-)Transkriptom untersuchen. Die vorhandenen Proteine analysieren wir in der Metaproteomik. Die Kombination dieser Beobachtungen ermöglicht es uns Stoffwechselprozesse vorherzusagen, wobei für die Hälfte der Proteine keine Funktion bekannt ist. Aber das gesammelte Wissen der Wissenschaft über bekannte Proteine und deren Funktion erlaubt uns einen ersten Blick auf neuartige Ökosysteme. Mit geschickten Experimenten, zum Beispiel bei der Beobachtung der Reaktion von Gemeinschaften auf Substratzugabe und der Untersuchung neuartiger Bakterienstämme, streben wir ein umfangreicheres Verständnis der wichtigsten Prozesse im Ökosystem Meer an. Wir sind immer noch Entdecker.



