- Abteilungen
- Abteilung Molekulare Ökologie
- Taxonomie
Taxonomie
Direktor
Geschäftsführender Direktor
MPI für Marine Mikrobiologie
Celsiusstr. 1
D-28359 Bremen
|
Raum: |
2221 |
|
Telefon: |

Unkultivierte Mikroben brauchen Taxonomie
Die Taxonomie umfasst die Identifizierung, Klassifizierung und Nomenklatur von Organismen. Als solche ist die Taxonomie eine Voraussetzung für die Ökologie. Nur auf der Grundlage genauer taxonomischer Konzepte und Methoden kann die Vielfalt und Zusammensetzung komplexer mikrobieller Gemeinschaften genau beschrieben und überwacht werden. Unsere Abteilung hat eine lange Tradition in der Entwicklung und Anwendung neuer taxonomischer Methoden. Wir haben Pionierarbeit bei der Identifizierung, Quantifizierung und Lokalisierung von noch nicht kultivierten Bakterien und Archaeen durch Fluoreszenz-in-situ-Hybridisierung (FISH) mit rRNA-gerichteten Oligonukleotid-Sonden geleistet (Amann et al. 1995; Amann & Fuchs 2008). Es besteht eine langjährige Zusammenarbeit mit Wolfgang Ludwig und Ralf Westram, den Hauptentwicklern des ARB-Programms, das weithin für die Rekonstruktion von 16S rRNA-basierten phylogenetischen Bäumen und für das Design von Oligonukleotid-Sonden verwendet wird. Hervorgegangen aus diesem Programm waren wir mit der Forschungsgruppe von Frank Oliver Glöckner auch lange die Heimat der viel genutzten, kuratierten 16S rRNA-Datenbank SILVA (Quast et al. 2013; Yilmaz et al. 2014). Diese ist nun an das Leibniz-Institut DSMZ-Deutsche Sammlung von Mikroorganismen und Zellkulturen übertragen worden.
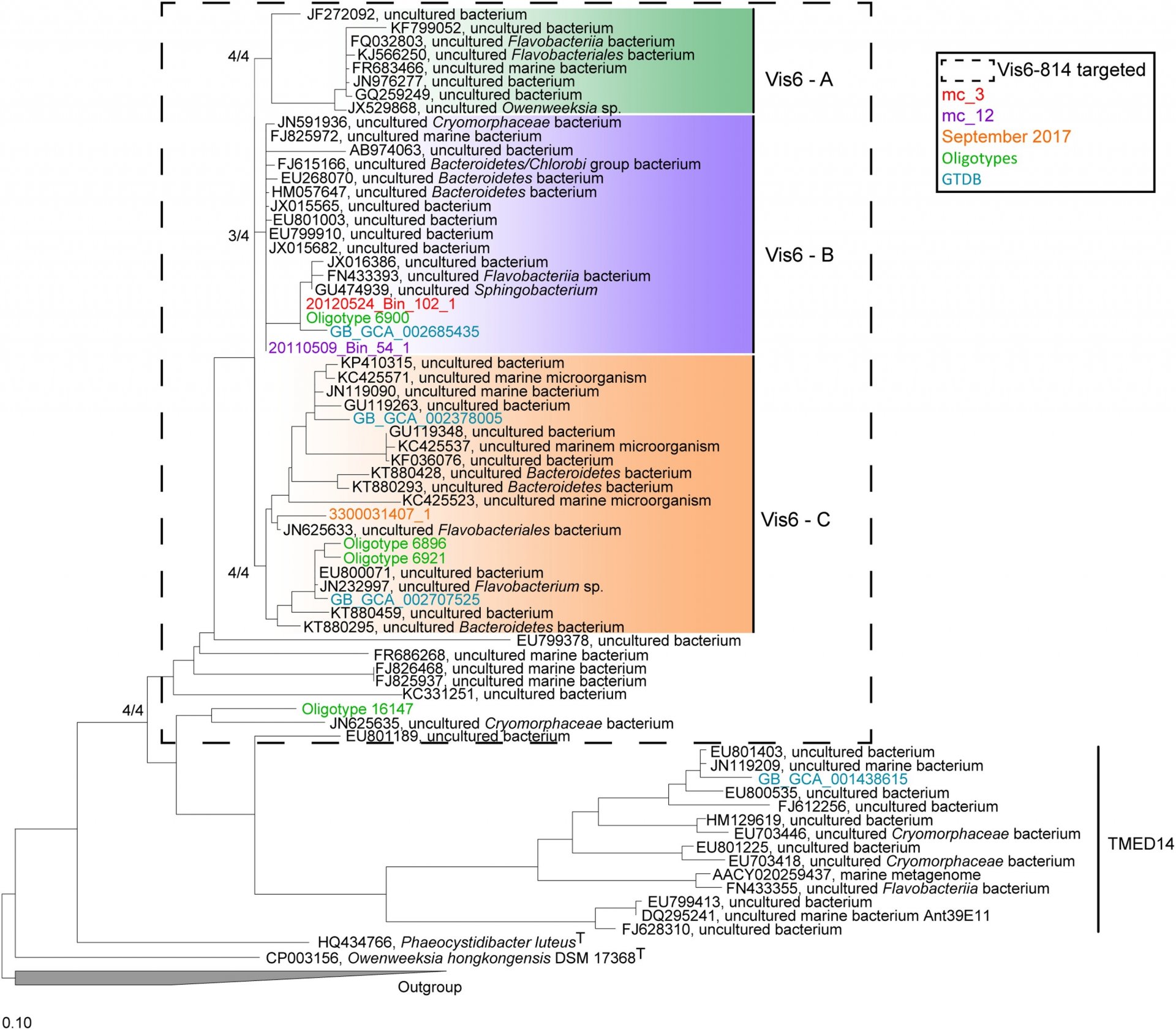
Da die meisten Mikroorganismen noch nicht kultiviert wurden, bestand ein Bedarf an Schwellenwerten, um zu beurteilen, ob ihre 16S rRNA-Fingerabdrücke auf die Existenz neuer Arten, Gattungen, Familien, Ordnungen, Klassen oder sogar neuer Phyla hinweisen. Wir haben zu weit verbreiteten Standards der 16S rRNA-basierten Klassifizierung beigetragen (Yarza et al. 2014). Heute ermöglicht die genomische Revolution sehr detaillierte, kultivierungsunabhängige Beschreibungen neuer Taxa, und es ist offensichtlich, dass Ordnung in deren unkontrollierte alphanumerische Benennung gebracht werden muss (Konstantinidis, Rosselló-Mora & Amann 2017). Die Einzelzellgenomik bzw. aus Metagenomen assemblierte Genome offenbaren uns, kombiniert mit Einzelzellidentifizierung durch FISH, die wahre Vielfalt der Mikroorganismen, die groß, aber sicher nicht unendlich ist (Amann & Rosselló-Mora 2016). Wir und andere haben die dringende Notwendigkeit erkannt, eine korrekte Klassifizierung der vielen funktionell wichtigen Gruppen von Umweltbakterien durchzuführen. Wir haben damit begonnen neue Taxa zu beschreiben, darunter die Art Candidatus Prosiliicoccus vernus (Francis et al. 2019) und die Gattung Candidatus Abditibacter (Grieb et al. 2020). Da der Rang von Candidatus jedoch vorläufig ist und die Nomenklatur keine Priorität hat, sind andere und wir davon überzeugt, dass genomische Informationen als Typusmaterial für die dauerhafte Beschreibung neuer Mikroorganismen akzeptiert werden müssen (Konstantinidis et al. 2020). Ökologen und Taxonomen benötigen einen Fahrplan für die Benennung von unkultivierten Archaeen und Bakterien (Murray et al. 2020), der den aktuellen Code of Nomenclature erweitert, der lebende Reinkulturen als Typusmaterial verlangt. Wir glauben, dass eine Taxonomie der noch nicht kultivierten Bakterien und Archaeen die Anreicherungs- und Kultivierungsarbeit nicht behindern wird, die mehr denn je benötigt wird.
Wir wissen jetzt nicht nur, dass viele „neue“ Mikroorganismen im Meer sind, sondern können auch vorhersagen, was sie dort tun. So können wir schon ökologische Zusammenhänge charakterisieren, während wir auf den Erfolg der Kultivierungsbemühungen warten, die dann noch genauere Untersuchung ermöglichen. Wir wissen viel über diese „Geister“ und sollten ihnen auf der Basis der Genome aussagekräftige Namen geben.