- Abteilungen
- Abteilung Molekulare Ökologie
- Forschungsgruppe Durchflusszytometrie
- Forschungsgebiete
Forschungsgebiete
Wachstums- und Sterblichkeitsraten von Bakterien
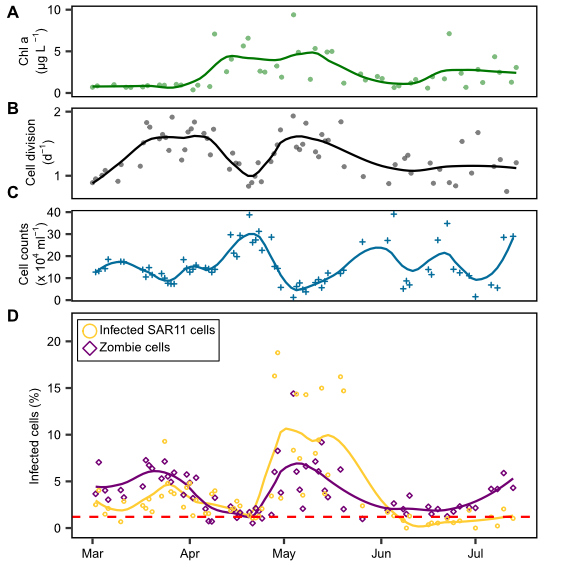
Wachstum ist ein grundlegender Parameter in der mikrobiellen Ökologie. Normalerweise werden die Wachstumsraten mit Hilfe von Verdünnungskulturen oder in Aufnahmeexperimenten mit radioaktiv markierten Substraten bestimmt. Jan Brüwer wählte in seiner Doktorarbeit einen zytometrischen Ansatz. Er suchte nach sich teilende Zellen, indem er die DNA-Verteilung innerhalb einzelner Zellen untersuchte. Zwei Maxima pro Zelle deuten auf eine sich teilende Zelle hin, kurz bevor sich die Zelle in zwei Tochterzellen aufteilt. Er kalibrierte diesen Ansatz erfolgreich mit Verdünnungskulturen und wandte ihn auf blühende Bakteriengruppen während zweier mariner Frühjahrsblüten in der Nordsee bei Helgoland an. Für copiotrophe Bakteriengruppen ermittelte er Zellteilungsraten von bis zu zweimal pro Tag (Brüwer et al., 2023). Überraschenderweise zeigte die SAR11 Gruppe während der Frühjahrsblüte Phasen extensiven Wachstums, ähnlich wie typische copiotrophe Kladen, gleichzeitig gingen aber ihre Zellzahlen zurück. Jan vermutete eine starke Sterblichkeit, die durch eine Phageninfektion verursacht wurde. Mithilfe von direct-geneFISH, das auf die drei wichtigsten Pelagiphagenspezies abzielt, zeigte er, dass während einer Phase mit hohem Wachstum und Zellrückgang bis zu 19% der SAR11-Zellen mit Phagen infiziert waren. Im globalen Durchschnitt waren zu jeder Zeit mehrere Prozent der SAR11-Zellen mit Phagen infiziert.
Während dieser Studie entdeckte Jan auch einen Zelltyp, in dem er die infizierenden Pelagiphagen hybridisieren konnte, die 16S ribosomale RNA aber nicht nachweisbar war. Er postulierte, dass sich diese Zellen in einem Übergangszustand mit hoher Phagenproduktion befinden, in denen sie hoch infiziert sind, aber noch nicht lysieren und noch keine neue Phagen-Nachkommen freisetzen. In diesem Stadium bauen die Viren möglicherweise ihre gesamte RNA ab, um die Nukleotide als Ressource für die Synthese neuer Phagengenome zu nutzen (Brüwer et al., 2024). Jan nannte Zellen in diesem Stadium „Zombiezellen“.
Zellsortierung für Proteomik Analysen
Die Vorhersage von metabolischen Funktionen aufgrund von (meta-)genomischer Annotation ist begrenzt. Ein Schritt näher am Metabolismus und der ökologischen Rolle eines Mikroorganismus sind seine exprimierten Proteine.
In diesem Projekt entwickeln wir die Kombination aus FISH und FACS weiter und kombinieren die Zellsortierung mit der Metaproteomik. In Pilotstudien konnten wir bereits mehrere hundert Peptide aus nur einer Million sortierter Zellen nach FISH identifizieren. Dieses Projekt ist Teil der DFG Forschungsgruppe FOR 2406 (Proteogenomics Of Marine Polysaccharide Utilization - POMPU).
>>Hier<< finden Sie mehr Informationen zum Unterprojekt 2: In situ mechanisms of polysaccharide degradation of key bacteroidetal genera in spring algae blooms.
Auf diesem Projekt arbeitet Grace Ho im Rahmen ihrer Doktorarbeit.
Visualisierung von zellulären Merkmalen in komplexen Planktonproben
In den letzten Jahren wurde das geneFISH-Protokoll in unserer Abteilung weiter optimiert, um spezifische Gene in komplexen natürlichen Proben nachzuweisen (Barrero-Canosa et al., 2017). Während ihrer Doktorarbeit gelang es Laura Zeugner, das Protokoll weiter zu vereinfachen, ohne Abstriche bei der Spezifität machen zu müssen. Innerhalb von 4 Stunden Inkubation konnte sie mit hochauflösender Mikroskopie Gene für zwei verschiedene Enzyme der Glykosidhydrolase-Familie 92 (GH92) in komplexen Planktonproben nachweisen. Beide GH92 bauen mannosehaltige Polysaccharide ab und werden zusammen mit anderen kohlenhydrataktiven Enzymen in sogenannten Polysaccharid-Utilization-Loci (PULs) kodiert. Laura konnte das GH92 eines konservierten PULs in einzelligen Polaribacter und Formosa in Meeresplanktonproben nachweisen. Interessanterweise entdeckte Laura nicht nur freilebende, sondern auch GH92-positive Zellen, die an Meerespartikel gebunden sind, was unsere ökologische Sicht auf den flavobakteriellen Mannoseabbau erweitert (Zeugner et al., 2021).

In ihrer Doktorarbeit verfolgte Greta Giljan einen weiteren, neuen Ansatz. Sie verwendete fluoreszierend-markierte Polysaccharide wie Laminarin und Fucoidan, um zu testen, welche Mikroorganismen diese Substrate über den "egoistischen" ("selfish") Mechanismus aufnehmen. In Kurzzeitinkubationen mit Helgoländer Nordseewasser wurden Zellen, die große Mengen an Polysacchariden in ihr Periplasma aufnahmen, mittels Durchflusszytometrie aussortiert und taxonomisch identifiziert. Insgesamt stellten wir starke saisonale Schwankungen bei der Polysaccharidaufnahme fest. Die stärksten Fluoreszenzsignale kamen insbesondere von Zellen, die zur verrucomicrobiellen Familie der Pedosphaeraceae (Giljan et al., 2022)

Nischenanpassung der weit verbreiteten Flavobakteriengruppe NS5
Seit seiner Entdeckung Anfang 2000 wurde die Flavobakteriengruppe NS5 häufig in Bakterioplankton-Analysen gefunden, aber noch nicht näher eingehend charakterisiert. Während seiner Doktorarbeit untersuchte Taylor Priest die NS5-Gruppe auf globaler Ebene und konnte sechs Gattungen abgrenzen, darunter vier neue NS5-Gattungen, die 35 Artencluster umfassen. Anhand von Proben aus der FRAM-Straße (Priest et al., 2021) stellte er einen Metagenomik-Datensatz zusammen und rekonstruierte den Stoffwechsel der NS5-Gattungen. Durch sorgfältige bioinformatische Analysen konnte Taylor die verschiedenen ökologischen Nischen dieser Arten auf globaler Ebene eingrenzen (Priest et al., 2022). NS5_D ist zum Beispiel an höhere Temperaturen angepasst, während NS5_F auf kalte polare Gewässer beschränkt ist. Während erstere ein höheres Polysaccharid-Abbaupotenzial hat, weist letztere die höchste Anzahl an TonB-abhängigen Transportersystemen auf. Zusammenfassend zeigt Taylor überzeugend, dass die Nischenaufteilung von NS5 sowohl von biotischen als auch von abiotischen Faktoren abhängt, und es ist ihm gelungen, für jede NS5-Gattung eine Reihe von Nischen-bestimmenden Faktoren zu identifizieren.

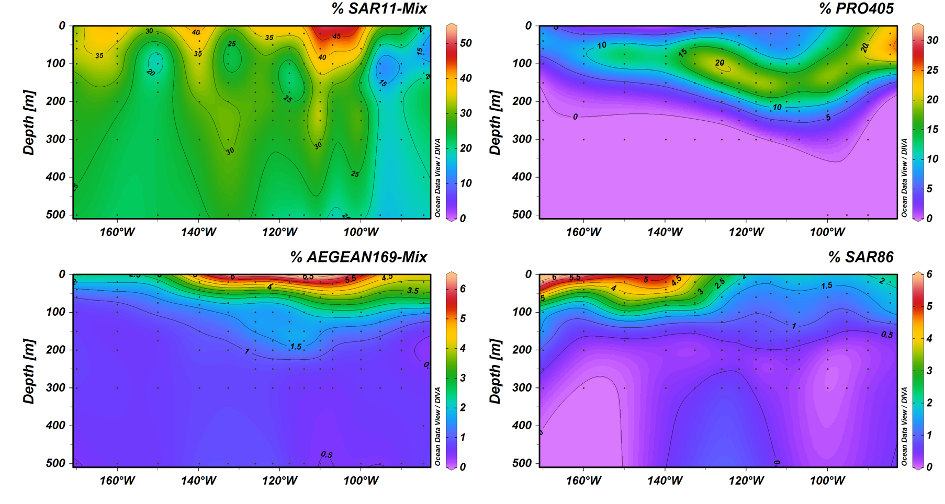
Südpazifischer Oceanwirbel
Der südpazifische Ozeanwirbel (SPG) ist die abgeschiedenste Meeresregion der Welt. In ihr befinden sich die klarsten Gewässer und findet sich die extremste Nährstofflimitation. Wegen dieser extremen Oligotrophie wird sie auch als „Ozeanwüste“ bezeichnet.
In einer gemeinsamen Forschungsreise mit der Abteilung für Biogeochemie fuhren wir durch den SPG, um die mikrobielle Gemeinschaft und ihre Fähigkeiten, mit solch extremen Bedingungen umzugehen, zu erforschen. Die Fahrt fand von Dezember 2015 bis Januar 2016 statt und führte quer durch den oligotrophen Südpazifikwirbel von Chile nach Neuseeland. Nach einem ersten Screening mit einer On-Board-Sequenzierungspipeline und On-Board-FISH-Analysen (Reintjes et al., 2019) konzentrierten wir uns auf die Charakterisierung neuer mikrobieller Gruppen durch gerichtete Durchflusszytometrie und Metagenomik. Neun Metagenome von drei Stationen und aus drei Oberflächenschichten wurden aus dem oligotrophen Zentrum des südpazifischen Wirbels erstellt und von Monika Oggerin eingehend analysiert.
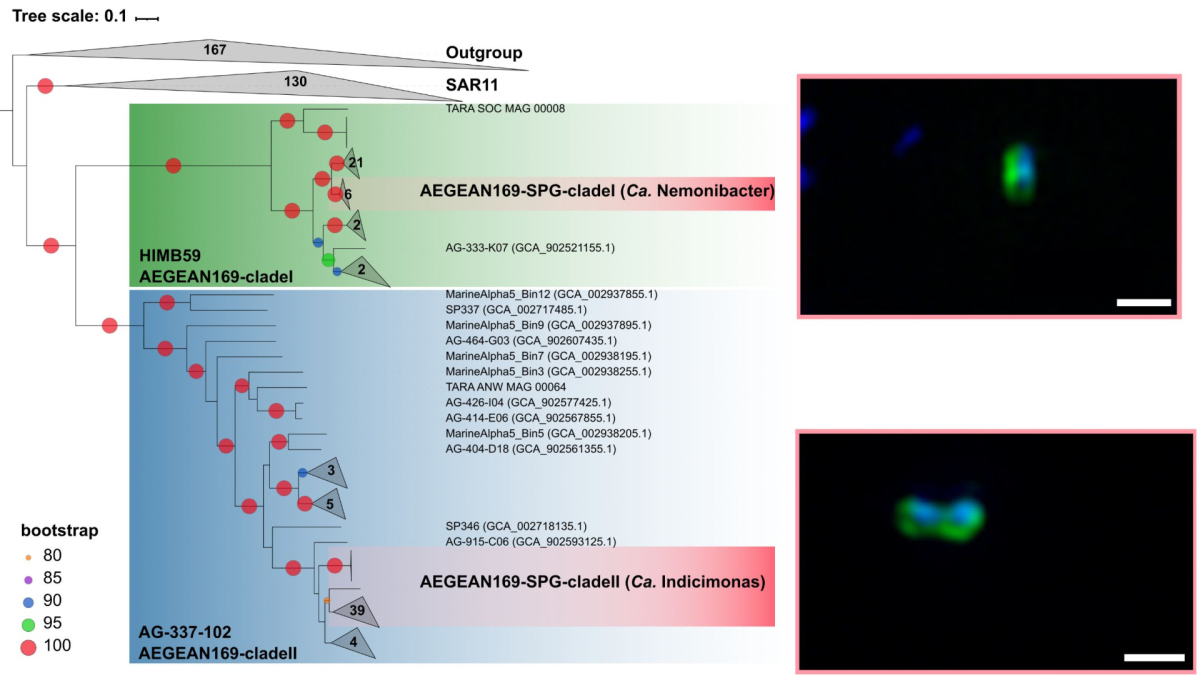
Eine der dominierenden Gruppen, die marine alphaproteobakterielle Gruppe AEGEAN169, wurde durch Fluoreszenz-in-situ-Hybridisierung mit relativen Häufigkeiten von bis zu 6% der gesamten mikrobiellen Gemeinschaft in der obersten Wasserschicht nachgewiesen. Innerhalb von AEGEAN169 gab es zwei verschiedene Genera, die Monika Candidatus Nemonibacter und Candidatus Indicimonas nannte (Oggerin et al., 2024 in Revision). Vergleichende metagenomische und metatranskriptomische Analysen der beiden AEGEAN169-Genera zeigten, dass sie im Vergleich zu ihren Konkurrenten SAR11, SAR86, SAR116 und Prochlorococcus subtile, aber unterschiedliche metabolische Anpassungen an die extremen Bedingungen im Südpazifik aufweisen. Beide AEGEAN169-Genera wiesen den höchsten Prozentsatz an Transportern pro vorhergesagtem Protein auf (9,5 bzw. 10,6%), der bisher für Bakterien berichtet wurde. Vor allem ABC-Transporter wurden in hohem Maße exprimiert, was in Kombination mit den Proteorhodopsinen darauf hindeutet, dass beide AEGEAN169-Genera von einer Vielzahl an freien Substraten leben können. Zusammenfassend lässt sich sagen, dass beide Genera in einzigartiger Weise an die knappen Ressourcen in den obersten Schichten des Südpazifiks angepasst sind und in hohem Maße konkurrenzfähig sind (Oggerin et al., 2024, in Revision).