Seitenpfad:
- Presse
- Pressemeldungen 2015
- 02.09.2015 Von der Natur lernen
02.09.2015 Von der Natur lernen
Von der Natur lernen: Gen-Standard erleichtert die Suche nach neuen natürlichen Wirkstoffen
Penicillin und andere sekundäre Metaboliten
Bremen, 2. September 2015
Jeder kennt Penicillin, ein wirksames Antibiotikum, das Alexander Fleming 1928 durch Zufall entdeckte. Penicillin ist ein sogenannter sekundärer Metabolit aus einem Schimmelpilz, der das Wachstum von Bakterien hemmt. Inzwischen verlässt man sich nicht mehr auf Zufallsfunde, denn mit systematischer Forschung hat man viele weitere dieser sekundären Metabolite, wie das antibakterielle Medikament Erythromycin, identifiziert. Die immense Bedeutung der Naturstoffe für Medizin, Landwirtschaft und Biotechnologie ist unbestritten. Viele Lebewesen produzieren diese kleinen exotischen Moleküle über mehrere Synthesestufen, und die Wissenschaft sucht mittlerweile mit computergestützten Methoden nach neuen Stoffen und deren Anwendungen. Jetzt hat ein internationales Konsortium von Forschern die zukünftigen Minimal-Standards für die Datenbanken in der Fachzeitschrift Nature Chemical Biology publiziert, um die Suche nach neuen Wirkstoffen voranzutreiben.
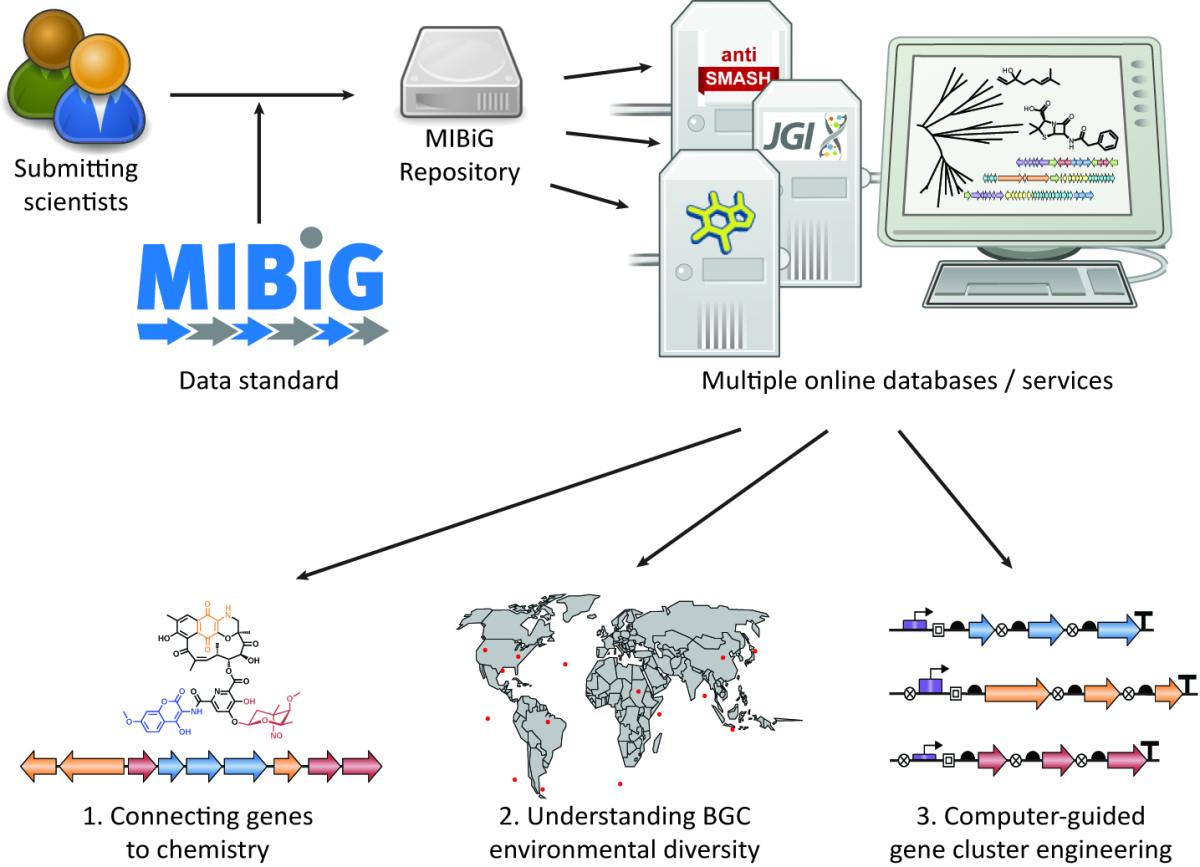
Der Ansatz über die Analyse der Gen-Daten verspricht Zeit- und Kostenersparnis, denn die für die Synthese verantwortlichen Gen-Gruppen, so genannte Biosynthetische Gen-Cluster (BGC), lassen sich aus der Erbsubstanz ableiten, erst im zweiten Schritt kommt der aufwendige experimentelle Nachweis. Vor über zehn Jahren schon formulierte das Genomic Standard Consortium (GSC) die ersten Standards für Gen-Datenbanken.
Prof. Frank Oliver Glöckner leitet die Forschungsgruppe Mikrobielle Genomik und Bioinformatik am Bremer Max-Planck-Institut für Marine Mikrobiologie und hat dieses Konsortium zusammen mit Prof. Marnix Medema ins Leben gerufen. Er sagt: „Gegenwärtig sind die Informationen über diese Gen-Cluster noch weit verstreut in der Literatur. Ohne gemeinsamen Standard bleibt es sehr mühselig, diese Informationen zusammenzuführen und auszuwerten. Das Konsortium besteht aus einer Gruppe von mehreren hundert Forschern. Wir haben uns auf vier Parameter geeinigt, die für jeden Cluster hinterlegt sein müssen. Das sind im Prinzip die klassischen W-Fragen. Wer hat wo publiziert, welcher Gen-Ort in welchem Organismus, welcher Stoff wird produziert und wie wurde das nachgewiesen. Diese „Minimal Information about a Biosynthetic Gene Cluster“, kurz MIBiG, vernetzt die bestehenden Datenbanken und wird die Suche nach noch unbekannten Stoffen extrem beschleunigen.“
Zukünftig wird es möglich sein, mit Hilfe der verschiedenen Online-Datenbanken die Beziehungen zwischen den Gen-Clustern, ihren chemischen Synthesevermögen und ihrer biologischen Vielfalt besser zu verstehen.
Penicillin und andere sekundäre Metaboliten
Bremen, 2. September 2015
Jeder kennt Penicillin, ein wirksames Antibiotikum, das Alexander Fleming 1928 durch Zufall entdeckte. Penicillin ist ein sogenannter sekundärer Metabolit aus einem Schimmelpilz, der das Wachstum von Bakterien hemmt. Inzwischen verlässt man sich nicht mehr auf Zufallsfunde, denn mit systematischer Forschung hat man viele weitere dieser sekundären Metabolite, wie das antibakterielle Medikament Erythromycin, identifiziert. Die immense Bedeutung der Naturstoffe für Medizin, Landwirtschaft und Biotechnologie ist unbestritten. Viele Lebewesen produzieren diese kleinen exotischen Moleküle über mehrere Synthesestufen, und die Wissenschaft sucht mittlerweile mit computergestützten Methoden nach neuen Stoffen und deren Anwendungen. Jetzt hat ein internationales Konsortium von Forschern die zukünftigen Minimal-Standards für die Datenbanken in der Fachzeitschrift Nature Chemical Biology publiziert, um die Suche nach neuen Wirkstoffen voranzutreiben.
Der Ansatz über die Analyse der Gen-Daten verspricht Zeit- und Kostenersparnis, denn die für die Synthese verantwortlichen Gen-Gruppen, so genannte Biosynthetische Gen-Cluster (BGC), lassen sich aus der Erbsubstanz ableiten, erst im zweiten Schritt kommt der aufwendige experimentelle Nachweis. Vor über zehn Jahren schon formulierte das Genomic Standard Consortium (GSC) die ersten Standards für Gen-Datenbanken.
Prof. Frank Oliver Glöckner leitet die Forschungsgruppe Mikrobielle Genomik und Bioinformatik am Bremer Max-Planck-Institut für Marine Mikrobiologie und hat dieses Konsortium zusammen mit Prof. Marnix Medema ins Leben gerufen. Er sagt: „Gegenwärtig sind die Informationen über diese Gen-Cluster noch weit verstreut in der Literatur. Ohne gemeinsamen Standard bleibt es sehr mühselig, diese Informationen zusammenzuführen und auszuwerten. Das Konsortium besteht aus einer Gruppe von mehreren hundert Forschern. Wir haben uns auf vier Parameter geeinigt, die für jeden Cluster hinterlegt sein müssen. Das sind im Prinzip die klassischen W-Fragen. Wer hat wo publiziert, welcher Gen-Ort in welchem Organismus, welcher Stoff wird produziert und wie wurde das nachgewiesen. Diese „Minimal Information about a Biosynthetic Gene Cluster“, kurz MIBiG, vernetzt die bestehenden Datenbanken und wird die Suche nach noch unbekannten Stoffen extrem beschleunigen.“
Zukünftig wird es möglich sein, mit Hilfe der verschiedenen Online-Datenbanken die Beziehungen zwischen den Gen-Clustern, ihren chemischen Synthesevermögen und ihrer biologischen Vielfalt besser zu verstehen.
Rückfragen an
Prof. Dr. Frank Oliver Glöckner
Max-Planck-Institut für Marine Mikrobiologie, Celsiusstr. 1, D-28359 Bremen,
Telefon:0421 2028 – 970, [Bitte aktivieren Sie Javascript]
und an den Pressesprecher
Dr. Manfred Schlösser
Max-Planck-Institut für Marine Mikrobiologie, Celsiusstr. 1
D-28359 Bremen, Telefon:0421 2028 - 704
[Bitte aktivieren Sie Javascript]
Originalarbeit
Minimum Information about a Biosynthetic Gene cluster. Marnix H Medema, Renzo Kottmann, Pelin Yilmaz et al. Nature Chemical Biology 11, 625–631 (2015) doi:10.1038/nchembio.1890
Der vollständige Artikel ist hier http://www.nature.com/nchembio/journal/v11/n9/full/nchembio.1890.html
Der oben zitierte Artikel unterliegt den Creative Commons Attribution- Non Commercial-ShareAlike 3.0 Unported License. http://creativecommons.org/licenses/by-nc-sa/3.0/.
Prof. Dr. Frank Oliver Glöckner
Max-Planck-Institut für Marine Mikrobiologie, Celsiusstr. 1, D-28359 Bremen,
Telefon:0421 2028 – 970, [Bitte aktivieren Sie Javascript]
und an den Pressesprecher
Dr. Manfred Schlösser
Max-Planck-Institut für Marine Mikrobiologie, Celsiusstr. 1
D-28359 Bremen, Telefon:0421 2028 - 704
[Bitte aktivieren Sie Javascript]
Originalarbeit
Minimum Information about a Biosynthetic Gene cluster. Marnix H Medema, Renzo Kottmann, Pelin Yilmaz et al. Nature Chemical Biology 11, 625–631 (2015) doi:10.1038/nchembio.1890
Der vollständige Artikel ist hier http://www.nature.com/nchembio/journal/v11/n9/full/nchembio.1890.html
Der oben zitierte Artikel unterliegt den Creative Commons Attribution- Non Commercial-ShareAlike 3.0 Unported License. http://creativecommons.org/licenses/by-nc-sa/3.0/.
Schema des Ablaufs. Die Forscher geben ihre Daten über ein Online-Formular ein. Von dort geht es automatisch weiter an mehrere Datenbanken, die miteinander vernetzt sind. Der Zugang ist öffentlich: http://mibig.secondarymetabolites.org/index.html