- Presse
- Pressemeldungen 2020
- Das geheime Leben der Mikroben: Schnappschuss von Molekülen in einer Tiefseesymbiose
Das geheime Leben der Mikroben: Schnappschuss von Molekülen in einer Tiefseesymbiose
Die Bakterien in unserer Umwelt sind oft schwer zu erforschen: Sie sind winzig klein und leben oft unter Bedingungen, die im Labor schwer nachzustellen sind – beispielsweise in der Tiefsee oder als Symbionten innerhalb eines tierischen Wirts (oder beides, wie die symbiotischen Tiefseebakterien in der nun vorliegenden Studie). Untersuchungen des Erbmaterials der Bakterien verraten uns, wozu die Mikroben theoretisch imstande sind. Was sie allerdings tatsächlich machen, geht daraus nicht hervor. Dazu erforschen Wissenschaftlerinnen und Wissenschaftler das sogenannte Metabolom der Bakterien: Das umfasst alles, was mit ihrem Stoffwechsel zu tun hat einschließlich der Metabolite – Zwischenprodukte des Stoffwechsels der Organismen, zum Beispiel Eiweiße, Zucker oder Fette.
Ein Team von Forschenden um Benedikt Geier und Manuel Liebeke vom Bremer Max-Planck-Institut für Marine Mikrobiologie haben nun eine Methode entwickelt, mit der man einzelne Bakterien identifizieren und gleichzeitig erkennen kann, welche Metabolite in der Zelle vorhanden sind. Mit der neuen Methode erforschen sie, wie Bakterien als symbiotische Untermieter in Tiefseemuscheln wohnen und überleben. Liebeke und sein Team analysierten Hunderte von Stoffwechselprodukten auf einer Fläche kleiner als ein Quadratmillimeter. So können sie erkennen, wie symbiotische Mikroben in ihrem Wirt leben und kommunizieren. „Wir machen quasi einen Schnappschuss der Bakterien bei der Arbeit – so, wie sie in ihrer natürlichen Umwelt, in diesem Fall innerhalb einer tierischen Zelle, aktiv sind“, so Liebeke. „Und das können wir in beeindruckender Auflösung von wenigen Mikrometern, etwa zehnmal dünner als ein menschliches Haar.“
Schockgefroren für den besten Schnappschuss: Nicht nur was, auch wer!
„Wir nutzen für unsere Analysen schockgefrorenes Muschelgewebe, das wir in gefrorenem Zustand hauchdünn schneiden“, erklärt Benedikt Geier. „Von diesen Schnitten machen wir mit einer besonderen Massenspektrometrie-Technik, dem MALDI-MS-Imaging, einen Schnappschuss der chemischen Verbindungen der Zellen. Wenn wir diesen Schnappschuss dann im Detail analysieren, können wir sehr viele verschiedene Metabolite auf kleinstem Raum erkennen.“ Sie geben Aufschluss darüber, welche Stoffe die Bakterien wozu nutzen und wie das Zusammenleben mit ihrem Muschelwirt funktioniert. Neben dem MALDI-MSI am Bremer Max-Planck-Institut nutzten Liebeke und sein Team dazu ein neuen MS-Imaging-Prototypen an der Justus-Liebig-Universität Gießen in enger Zusammenarbeit mit Professor Spengler, wodurch besonders hochauflösende Einblicke möglich wurden.
Korrekte Schlussfolgerungen aus den Bildern der Metabolite sind nur möglich, wenn auch bekannt ist, wer sie erzeugt oder nutzt. „Bislang konnten wir zwar die Stoffwechselprodukte messen“, erklärt Geier, „aber wussten nicht, ob und wenn ja welche Bakterien beteiligt sind.“ Um dieses Problem zu lösen, nutzten die Forschenden zusätzlich eine weitere Methode, die sogenannte Fluoreszenz-in situ-Hybridisierung oder kurz FISH, um einzelne Bakterienzellen in der jeweiligen Probe zu identifizieren. „Durch die Kombination mit FISH können wir unsere MALDI-MS-Bilder nun sinnvoll erklären und den Bakterien im Muschelgewebe zuordnen.“
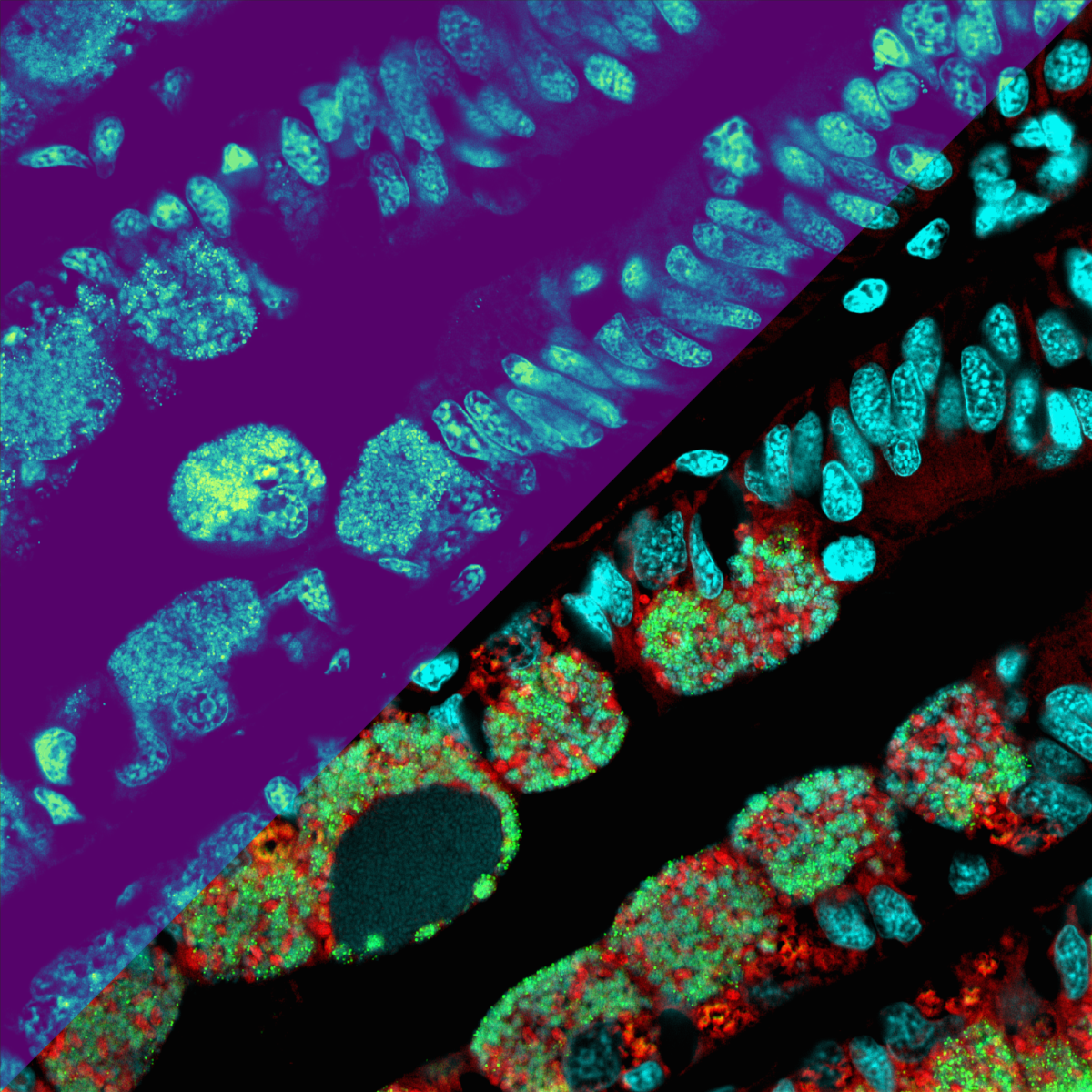
Aus der Tiefsee vor die Kamera
Die hier untersuchten Proben stammen von Schwarzen Rauchern in der Tiefsee – hochaufragenden Schloten, an denen heißes, mineralreiches Wasser aus dem Meeresgrund strömt. Tiere und Bakterien können dort nur in symbiotischer Gemeinschaft überleben. Geier untersucht in seiner Doktorarbeit das Zusammenleben von Bakterien und Muscheln, insbesondere die enge Vernetzung ihres Stoffwechsels. Dank der neuen Methode konnte er zeigen, dass sich die Zusammensetzung der Lipide in der Muschel in Körperregionen mit und ohne bakterielle Untermieter deutlich unterscheidet. „Bislang waren solche Einsichten nicht möglich, da die Proben vor der Analyse homogenisiert, also quasi zermatscht, wurden“, erklärt Geier. „Auch, dass unsere Methode an Proben direkt aus der Umwelt und nicht nur solchen aus dem Labor funktioniert, zeigt ihr großes Potenzial“, so Geier weiter.
Alle Pflanzen und Tiere und auch wir Menschen leben in Gemeinschaft mit Mikroorganismen, durch unsere eng vernetzten Stoffwechsel teilen wir Metabolite. „Die Anwendung dieser Methode auf andere Wirt-Mikroben-Gemeinschaften wird uns viele spannende neue Einsichten in das geheime Leben der Mikroben gewähren. Ich bin gespannt, ob wir dieses bildgebende Verfahren auch nutzen können, um die gemeinsame Chemie zwischen Mikroben und den Organen ganzer Tiere zu erforschen. Es gibt noch viel zu entdecken!“
Weiterführende Links
Preisgekrönte Methodik
Für diese innovative Methode erhielt Benedikt Geier den MSI Award, der alljährlich für herausragende wissenschaftliche Arbeiten mithilfe bildgebender Verfahren mit einem Massenspektrometer verliehen wird. Lesen Sie mehr dazu hier.
Originalveröffentlichung
Benedikt K. Geier, Emilia Sogin, Dolma Michellod, Moritz Janda, Mario Kompauer, Bernhard Spengler, Nicole Dubilier, Manuel Liebeke: Spatial metabolomics of in situ host-microbe interactions at the micrometre scale. Nature Microbiology.
Beteiligte Institutionen
- Max-Planck-Institut für Marine Mikrobiologie, Bremen
- MARUM, Zentrum für marine Umweltwissenschaften der Universität Bremen
- Justus-Liebig-Universität Gießen
Rückfragen bitte an:
Gruppenleiter
MPI für Marine Mikrobiologie
Celsiusstr. 1
D-28359 Bremen
|
Raum: |
3244 |
|
Telefon: |

Pressereferentin
MPI für Marine Mikrobiologie
Celsiusstr. 1
D-28359 Bremen
|
Raum: |
1345 |
|
Telefon: |


