Seitenpfad:
- Presse
- Pressemeldungen 2012
- 28.02.2012 Introns in bakterieller 16S rRNA gef...
28.02.2012 Introns in bakterieller 16S rRNA gefunden
Das Merkmal, mit dem die Mikrobiologen die stammesgeschichtliche Zugehörigkeit von Bakterien festlegen, muss bestimmte Ansprüche erfüllen. Konservativ in seiner Funktion muss es sein und in allen Lebewesen vorkommen. Diese Anforderungen erfüllt in besonders hohem Maße ein bestimmtes Gen: das Gen für die kleinere der beiden Untereinheiten, aus dem die Ribosomen aufgebaut sind, auch bekannt als 16S rRNA-Gen. Deshalb wird es besonders häufig für phylogenetische Analysen verwendet, mit denen die Forscher den Grad der Verwandtschaft bei Mikroben bestimmen.
Verena Salman vom Max-Planck-Institut für Marine Mikrobiologie in Bremen hat nun zusammen mit ihren Kollegen dieses 16S rRNA-Gen einer Bakteriengruppe, den großen Schwefelbakterien genauer untersucht (Abbildung 1). Sie haben herausgefunden, dass das 16S rRNA-Gen mancher Vertreter dieser Bakteriengruppe an bis zu vier Stellen durch lange DNA-Abschnitte unterbrochen ist (Abbildung 2). Bei diesen so genannten Introns handelt es sich allerdings nicht um Genabschnitte, die für die 16S rRNA kodieren: Teilweise bestehen sie einfach aus nichtkodierender DNA oder sie tragen Gene für bestimmte Enzyme, durch die sich das Intron in andere Stellen des Genoms ausbreiten kann.
Verena Salman vom Max-Planck-Institut für Marine Mikrobiologie in Bremen hat nun zusammen mit ihren Kollegen dieses 16S rRNA-Gen einer Bakteriengruppe, den großen Schwefelbakterien genauer untersucht (Abbildung 1). Sie haben herausgefunden, dass das 16S rRNA-Gen mancher Vertreter dieser Bakteriengruppe an bis zu vier Stellen durch lange DNA-Abschnitte unterbrochen ist (Abbildung 2). Bei diesen so genannten Introns handelt es sich allerdings nicht um Genabschnitte, die für die 16S rRNA kodieren: Teilweise bestehen sie einfach aus nichtkodierender DNA oder sie tragen Gene für bestimmte Enzyme, durch die sich das Intron in andere Stellen des Genoms ausbreiten kann.
Abbildung1: Die Zellen der großen Schwefelbakterien Thiomargarita leben oft in Ketten zusammen. In ihren 16SrRNA-Gen haben die Wissenschaftler vier Introns gefunden. Die Zellen sind mit einem Durchmesser von ca. 200 μm außergewöhnlich groß für Bakterienzellen. Bild: V. Salman
„Die Introns sind jeweils mehrere hundert Basenpaare lang. Dadurch verlängern sie das 16S rRNA-Gen deutlich. Wir haben bis zu 3500 Basenpaare lange 16S rRNA-Gene gefunden“, sagt Verena Salman. Das normale 16S rRNA-Gen ist nur etwa 1500 Basenpaare lang. Den Forschern gelang jetzt zum ersten Mal der Nachweis dieser Introns in dem Markergen der bakteriellen 16S rRNA. Dies ist erstaunlich, da dieses Gen das am häufigsten sequenzierte Gen ist.
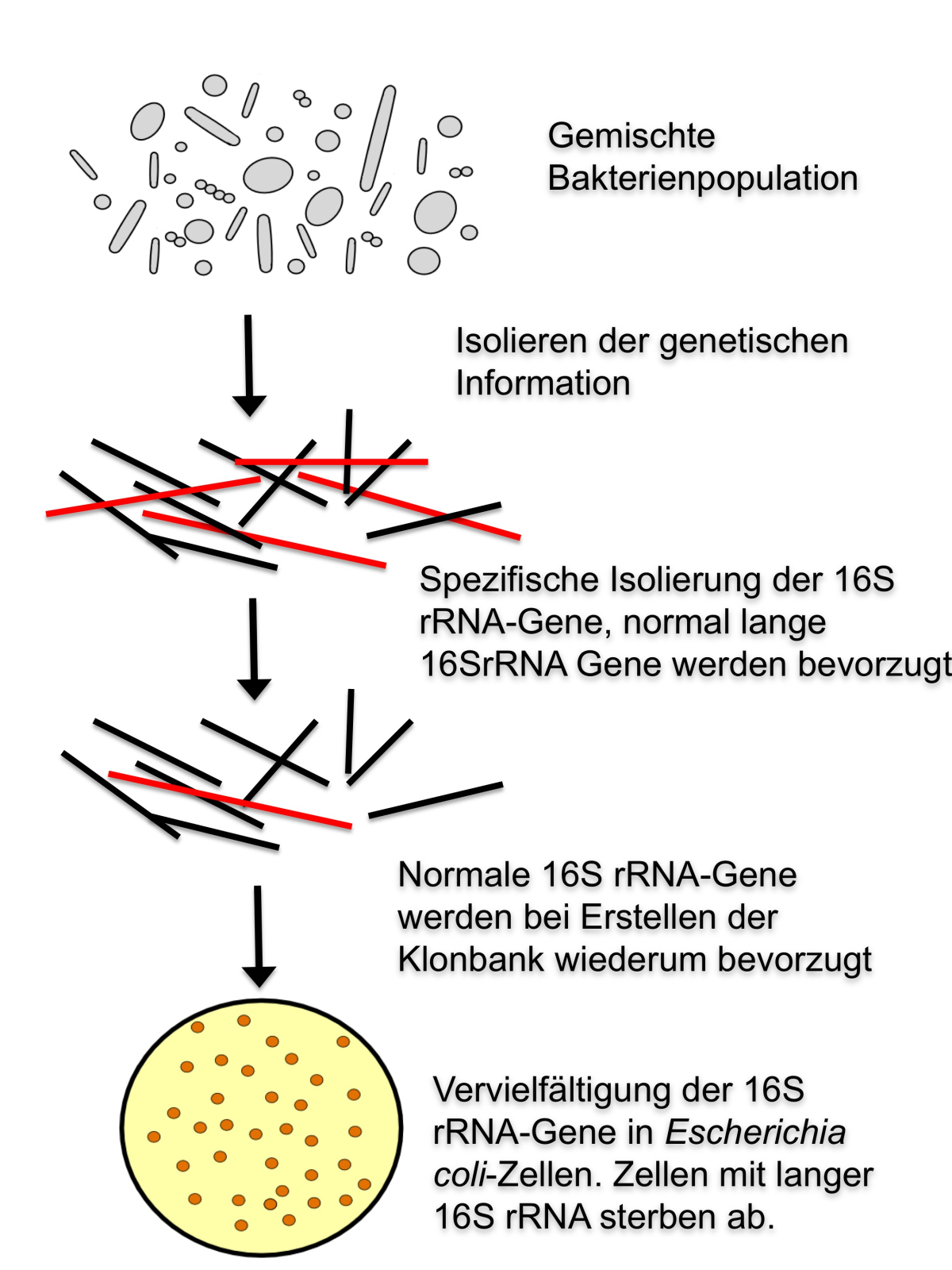
Für die Mikrobiologen hat die Entdeckung der Introns im 16S rRNA-Gen gravierende Auswirkungen. „Bisher konnten wir nur kurze Stücke der 16S rRNA-Gene der großen Schwefelbakterien aus einer gemischten Bakterienprobe sequenzieren. Nun wissen wir warum: Ihre 16S rRNA-Gene sind länger als die anderer Bakterien,“ erklärt Rudolf Amann, Direktor des Max-Planck-Instituts. „Mikrobenzellen, die diese Variante tragen, können mit den klassischen Nachweismethoden nicht gefunden werden, weil die kürzeren Sequenzen ungleich stärker ins Gewicht fallen.“ Deshalb gelang es den Wissenschaftlern bisher nicht, das Gen in seiner ganzen Länge nachzuweisen (Abbildung 3).
Für die Mikrobiologen hat die Entdeckung der Introns im 16S rRNA-Gen gravierende Auswirkungen. „Bisher konnten wir nur kurze Stücke der 16S rRNA-Gene der großen Schwefelbakterien aus einer gemischten Bakterienprobe sequenzieren. Nun wissen wir warum: Ihre 16S rRNA-Gene sind länger als die anderer Bakterien,“ erklärt Rudolf Amann, Direktor des Max-Planck-Instituts. „Mikrobenzellen, die diese Variante tragen, können mit den klassischen Nachweismethoden nicht gefunden werden, weil die kürzeren Sequenzen ungleich stärker ins Gewicht fallen.“ Deshalb gelang es den Wissenschaftlern bisher nicht, das Gen in seiner ganzen Länge nachzuweisen (Abbildung 3).
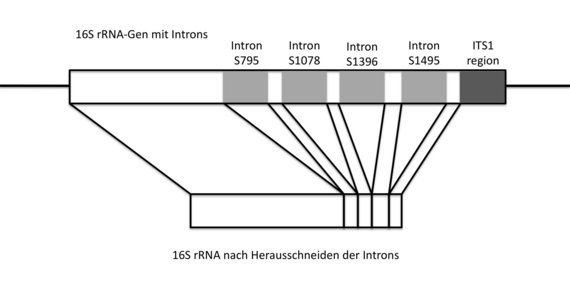
Abbildung 2: In der schematischen Darstellung sind die Positionen, an der die Introns auf dem 16S rRNA-Gen liegen, hellgrau markiert. Bevor das fertige Ribosom gebildet wird, werden die Introns herausgeschnitten. Abbildung: V. Salman/R. Dunker
Da das 16S rRNA-Gen eine wichtige Rolle bei der stammesgeschichtlichen Einordnung von Bakterien und in Biodiversitätsstudien spielt, hat die Entdeckung der Bremer Forscher weitreichende Folgen. Denn vermutlich gibt es die Introns auch in den 16S rRNA-Genen anderer Bakteriengruppen. „Wir versuchen jetzt herauszufinden, welche anderen Mikroorganismen auch das verlängerte 16S rRNA-Gen tragen,“ so Verena Salman. „Mit dem neuen Wissen werden wir eine Strategie entwickeln, um auch die langen 16S rRNA-Sequenzen zu berücksichtigen, beispielsweise bei Biodiversitätsstudien“.
Für die Bakterien haben die Introns in den 16S rRNA-Genen scheinbar keine Auswirkungen. Trotz der langen Introns bilden die Zellen der großen Schwefelbakterien funktionsfähige Ribosomen aus. Die Introns entfernen sich bei der Entstehung der Ribosomen selbst (Abbildung 2).
Für die Bakterien haben die Introns in den 16S rRNA-Genen scheinbar keine Auswirkungen. Trotz der langen Introns bilden die Zellen der großen Schwefelbakterien funktionsfähige Ribosomen aus. Die Introns entfernen sich bei der Entstehung der Ribosomen selbst (Abbildung 2).
Abbildung 3: 16S rRNA-Gene aus einer gemischten Bakterienpopulation können mit den Standardmethoden nur gefunden werden, wenn sie einer bestimmten Größenklasse zugehören. Längere 16S rRNA-Gene, z.B. solche mit Introns, werden bei dieser Standardmethode vernachlässigt und können daher bei regulären Biodiversitätsstudien nicht erfasst werden. Normal lange 16S rRNA-Gene sind schwarz, verlängerte rot markiert. Abbildung: V. Salman/R. Dunker
Rückfragen an
Dr. Verena Salman
Prof. Dr. Heide Schulz-Vogt
Prof. Dr. Rudolf Amann
Oder an die Pressesprecher
Dr. Rita Dunker
Dr. Manfred Schlösser
Originalarbeit
Multiple self-splicing introns in the 16S rRNA genes of giant sulfur bacteria, 2012. V. Salman, R. Amann, D. A. Shub, and H. Schulz-Vogt. Proceedings of the National Academy of Science, Early Edition.
DOI: 10.1073/pnas.1120192109
Beteiligte Institute
Max-Planck-Institut für Marine Mikrobiologie, Bremen
University of Albany, State University of New York, Albany, USA
Dr. Verena Salman
Prof. Dr. Heide Schulz-Vogt
Prof. Dr. Rudolf Amann
Oder an die Pressesprecher
Dr. Rita Dunker
Dr. Manfred Schlösser
Originalarbeit
Multiple self-splicing introns in the 16S rRNA genes of giant sulfur bacteria, 2012. V. Salman, R. Amann, D. A. Shub, and H. Schulz-Vogt. Proceedings of the National Academy of Science, Early Edition.
DOI: 10.1073/pnas.1120192109
Beteiligte Institute
Max-Planck-Institut für Marine Mikrobiologie, Bremen
University of Albany, State University of New York, Albany, USA