Seitenpfad:
- Presse
- Pressemeldungen 2011
- 12.05.2011 Genomdaten - Neue Standards
12.05.2011 Genomdaten - Neue Standards
Wie man mit wertvollen Genomdaten umgehen sollte.
MIMARKS und MIxS - neue Minimum-Informationsstandards für Marker-Gene und andere Sequenzen in der Umweltforschung
Ein Gruppe von fast 100 Wissenschaftlern des Genomic Standards Consortium (GSC) hat jetzt einen neuen Standard und einen Kriterienkatalog für die Umweltforschung vorgestellt. Entwickelt unter der Leitung von Forschern des Bremer Max-Planck-Instituts für Marine Mikrobiologie, des NERC Centre for Ecology and Hydrology (CEH) und der University of Colorado wird dieser zu einer erheblichen Verbesserung der Genom-, Metagenom- und Marker-Gen-Einträge führen. Zurzeit fehlen bei den meisten Sequenz-Datensätzen in den öffentlichen Datenbanken grundlegende Informationen über die jeweiligen Habitate und deren genaue geographische Positionen. Jetzt stellen die Forscher des GSC ihren MIMARKS-Standard (Minimum Information about a MARKer gene Sequence) als die neueste Checkliste als Teil der MIxS (Minimum Information about any (x) Sequence) Spezifikationen in dem angesehenen Fachblatt Nature Biotechnology vor (doi:10.1038/nbt.1823).
MIMARKS und MIxS - neue Minimum-Informationsstandards für Marker-Gene und andere Sequenzen in der Umweltforschung
Ein Gruppe von fast 100 Wissenschaftlern des Genomic Standards Consortium (GSC) hat jetzt einen neuen Standard und einen Kriterienkatalog für die Umweltforschung vorgestellt. Entwickelt unter der Leitung von Forschern des Bremer Max-Planck-Instituts für Marine Mikrobiologie, des NERC Centre for Ecology and Hydrology (CEH) und der University of Colorado wird dieser zu einer erheblichen Verbesserung der Genom-, Metagenom- und Marker-Gen-Einträge führen. Zurzeit fehlen bei den meisten Sequenz-Datensätzen in den öffentlichen Datenbanken grundlegende Informationen über die jeweiligen Habitate und deren genaue geographische Positionen. Jetzt stellen die Forscher des GSC ihren MIMARKS-Standard (Minimum Information about a MARKer gene Sequence) als die neueste Checkliste als Teil der MIxS (Minimum Information about any (x) Sequence) Spezifikationen in dem angesehenen Fachblatt Nature Biotechnology vor (doi:10.1038/nbt.1823).
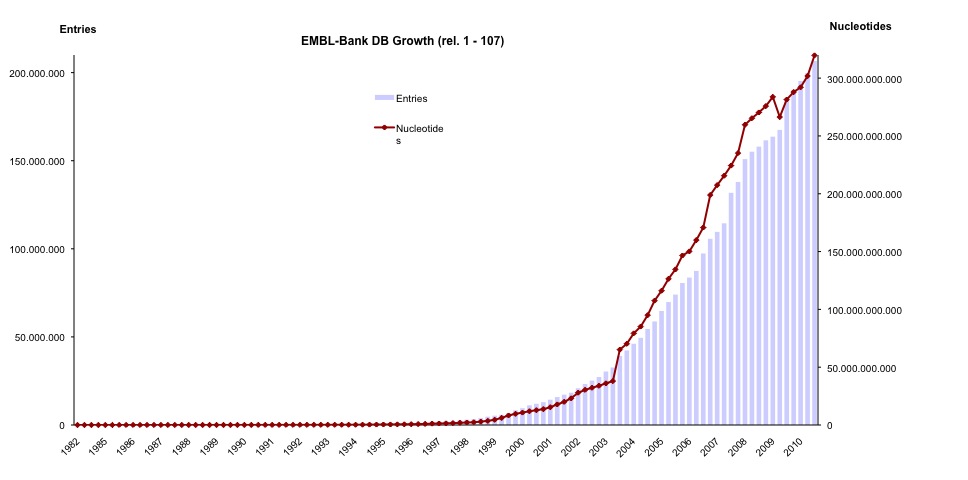
Diese Graph verdeutlicht das exponentielle Anwachsen der Einträge in die öffentlichen Datenbanken (INDSC) in den letzten dreißig Jahren.
Das Tempo, mit dem in Sequenzierungsprojekten neue Daten generiert werden, ist in letzter Zeit dramatisch angestiegen. Die Entwicklung neuer Hochdurchsatz-Technologien führte dazu, dass in der Wissenschaft Mega-Sequenzierungs-Projekte möglich wurden, die sich mit der Analyse von Organismen in so verschiedenen Habitaten wie dem Boden, den Meeren und sogar dem menschlichen Körper befassen. Die Wissenschaftler sammeln Proben und stellen die entsprechenden Sequenzdaten in die öffentlich zugänglichen Datenbanken wie dem European Nucleotide Archive, GenBank und der DNA Data Bank of Japan, die sich zur International Nucleotide Sequence Database Collaboration (INDSC) zusammengeschlossen haben. Leider fehlen in diesen Datenbanken meistens wichtige Meta-Daten wie die geographische Lage, den genauen Zeitpunkt der Probenahme, die Art des Habitats sowie weitere wichtig Parameter. Diese Daten können später nur mühevoll nachgearbeitet werden, in dem die Originalliteratur herangezogen, oder bei den Autoren direkt nachgefragt werden muss, um dann möglicherweise festzustellen,dass diese Information nicht mehr existiert.
Prof. Dr. Frank Oliver Glöckner vom Bremer Max-Planck-Institut für Marine Mikrobiologie und der Jacobs University Bremen sagt: “Sequenzinformationen ohne exakte Hintergrunddaten sind wie neue technische Gerätschaften ohne Handbuch. Man kann die jeweiligen Funktionen zwar erraten, aber ein effektiver Einsatz ist doch unwahrscheinlich. Wir hoffen, dass wir mit der neuen MIMARKS-Checkliste und den MIxS-Spezifikationen diese Art Probleme aus der Welt schaffen können und damit der Wissenschaft neue Wege der Meta-Analyse von Sequenzdaten erschließen“.
Den Autoren der MIMARKS- und MIxS-Projekte war von Anfang an klar, dass diese Wege nur beschritten werden können, wenn alle anderen Wissenschaftler mitmachen und ihre Datensätze einheitlich, entsprechend Inhalt, Syntax und Terminologie, strukturieren. Die Initiative begann im Jahr 2005, als sich die Forscher aus verschiedenen Bereichen zum ersten Mal trafen und das Genomic Standards Consortium (GSC) gründeten, eine für jeden offenen Gruppe mit dem Ziel, die Parameter für die Genome, Metagenome und zugehöriger Daten zu verbessern (Field D et al. (2008) The minimum information about a genome sequence (MIGS) specification. Nature Biotechnology 26:541-547). Nach zwei weiteren Jahren intensiven Austauschs veröffentlichen sie jetzt die MIMARKS-Checkliste und die MIxS-Spezifikationen im Internet unter http://www.gensc.org/gc_wiki/index.php/MIMARKS und http://www.gensc.org/gc_wiki/index.php/MIxS.
Diese Initiative findet bereits jetzt großen Anklang in der Wissenschaft, und ist getragen durch Forscher aus vielen Bereichen. Die Palette reicht von führenden Wissenschaftlern, großen wissenschaftliche Vereinigungen, Sequenzier-Zentren bis hin zu Forschern, die noch am Anfang ihrer Karriere stehen. Denn es geht um nichts geringeres, als den freien und offenen Austausch von wichtigen Forschungsergebnissen zu garantieren.
“Diese neuen Regeln werden das Leben zukünftiger Forscher-generationen einfacher machen”, sagt Dr. Renzo Kottmann vom Bremer Max-Planck-Institut. Dr. Rob Knight von der University of Colorado ergänzt: ”Jeder Forscher wird zukünftig schnelle und präzise Antworten auf grundlegende Fragen finden: Wurden meine Mikroben schon woanders gesichtet, und falls ja, wo, mit wem unter welchen besonderen Umweltbedingungen?“.
Prof. Dawn Field vom CEH führt weiter aus: “Der MIMARKS-Standard baut auf die anderen etablierten Genomstandards MIGS und MIMS auf. Damit haben wir eine Familie von Standards für die Bereiche Genom, Metagenom und Gen-Marker-Sequenzen geschaffen. Diese Publikation ist der Abschluss langjähriger Arbeit vieler Wissenschafter und hoffentlich der Anfang einer neuen Ära“.
Manfred Schlösser
Für weitere Informationen wenden Sie sich bitte an
Prof. Frank Oliver Glöckner und Pelin Yilmaz
oder an den Pressesprecher Manfred Schlösser
unter [Bitte aktivieren Sie Javascript]
Originalartikel:
Minimum information about a marker gene sequence (MIMARKS) and minimum information about any (x) sequence (MIxS) specifications
Pelin Yilmaz, Renzo Kottmann, Dawn Field, Rob Knight, Frank Oliver Glöckner and around 100 other scientists. NATURE Biotechnology 2011. (doi:10.1038/nbt.1823)
and
The genomic standards consortium: bringing standards to life for microbial ecology
Pelin Yilmaz, Jack A Gilbert, Rob Knight, Linda Amaral-Zettler, Ilene Karsch-Mizrachi, Guy Cochrane, Yasukazu Nakamura, Susanna-Assunta Sansone, Frank Oliver Glöckner and Dawn Field
doi:10.1038/ismej.2011.39, http://www.nature.com/ismej/journal/vaop/ncurrent/full/ismej201139a.html
Prof. Dr. Frank Oliver Glöckner vom Bremer Max-Planck-Institut für Marine Mikrobiologie und der Jacobs University Bremen sagt: “Sequenzinformationen ohne exakte Hintergrunddaten sind wie neue technische Gerätschaften ohne Handbuch. Man kann die jeweiligen Funktionen zwar erraten, aber ein effektiver Einsatz ist doch unwahrscheinlich. Wir hoffen, dass wir mit der neuen MIMARKS-Checkliste und den MIxS-Spezifikationen diese Art Probleme aus der Welt schaffen können und damit der Wissenschaft neue Wege der Meta-Analyse von Sequenzdaten erschließen“.
Den Autoren der MIMARKS- und MIxS-Projekte war von Anfang an klar, dass diese Wege nur beschritten werden können, wenn alle anderen Wissenschaftler mitmachen und ihre Datensätze einheitlich, entsprechend Inhalt, Syntax und Terminologie, strukturieren. Die Initiative begann im Jahr 2005, als sich die Forscher aus verschiedenen Bereichen zum ersten Mal trafen und das Genomic Standards Consortium (GSC) gründeten, eine für jeden offenen Gruppe mit dem Ziel, die Parameter für die Genome, Metagenome und zugehöriger Daten zu verbessern (Field D et al. (2008) The minimum information about a genome sequence (MIGS) specification. Nature Biotechnology 26:541-547). Nach zwei weiteren Jahren intensiven Austauschs veröffentlichen sie jetzt die MIMARKS-Checkliste und die MIxS-Spezifikationen im Internet unter http://www.gensc.org/gc_wiki/index.php/MIMARKS und http://www.gensc.org/gc_wiki/index.php/MIxS.
Diese Initiative findet bereits jetzt großen Anklang in der Wissenschaft, und ist getragen durch Forscher aus vielen Bereichen. Die Palette reicht von führenden Wissenschaftlern, großen wissenschaftliche Vereinigungen, Sequenzier-Zentren bis hin zu Forschern, die noch am Anfang ihrer Karriere stehen. Denn es geht um nichts geringeres, als den freien und offenen Austausch von wichtigen Forschungsergebnissen zu garantieren.
“Diese neuen Regeln werden das Leben zukünftiger Forscher-generationen einfacher machen”, sagt Dr. Renzo Kottmann vom Bremer Max-Planck-Institut. Dr. Rob Knight von der University of Colorado ergänzt: ”Jeder Forscher wird zukünftig schnelle und präzise Antworten auf grundlegende Fragen finden: Wurden meine Mikroben schon woanders gesichtet, und falls ja, wo, mit wem unter welchen besonderen Umweltbedingungen?“.
Prof. Dawn Field vom CEH führt weiter aus: “Der MIMARKS-Standard baut auf die anderen etablierten Genomstandards MIGS und MIMS auf. Damit haben wir eine Familie von Standards für die Bereiche Genom, Metagenom und Gen-Marker-Sequenzen geschaffen. Diese Publikation ist der Abschluss langjähriger Arbeit vieler Wissenschafter und hoffentlich der Anfang einer neuen Ära“.
Manfred Schlösser
Für weitere Informationen wenden Sie sich bitte an
Prof. Frank Oliver Glöckner und Pelin Yilmaz
oder an den Pressesprecher Manfred Schlösser
unter [Bitte aktivieren Sie Javascript]
Originalartikel:
Minimum information about a marker gene sequence (MIMARKS) and minimum information about any (x) sequence (MIxS) specifications
Pelin Yilmaz, Renzo Kottmann, Dawn Field, Rob Knight, Frank Oliver Glöckner and around 100 other scientists. NATURE Biotechnology 2011. (doi:10.1038/nbt.1823)
and
The genomic standards consortium: bringing standards to life for microbial ecology
Pelin Yilmaz, Jack A Gilbert, Rob Knight, Linda Amaral-Zettler, Ilene Karsch-Mizrachi, Guy Cochrane, Yasukazu Nakamura, Susanna-Assunta Sansone, Frank Oliver Glöckner and Dawn Field
doi:10.1038/ismej.2011.39, http://www.nature.com/ismej/journal/vaop/ncurrent/full/ismej201139a.html